杨若林等《Journal of Genetics and Genomics》2022年
作者: 来源:伟德BETVLCTOR1946 发布日期:2022-07-18 浏览次数:
论文题目:Deciphering the regulatory code of histone modifications in plants
论文作者:ZhaohongLi,DongweiLi,YeLi,XiaopingGuo,RuolinYang
论文摘要:该研究首先利用随机森林回归机器学习模型对拟南芥、水稻和玉米三种植物多种组织的19种组蛋白修饰ChIP-seq数据进行了建模,证明仅从DNA序列特征(k-mers)也能较准确地预测出植物组蛋白修饰水平,并利用该算法发掘和鉴定出一千多个具有潜在调控组蛋白修饰功能的非冗余DNA基序(motif),其中部分基序与已知参与调控植物组蛋白修饰的转录因子DNA基序吻合。进化分析表明,鉴定到的DNA基序总体受到较强的功能约束,进化速率较小,提示它们在组蛋白修饰中具有重要功能。综上,该论文解析了植物组蛋白修饰的DNA调控语法,为其动态进化模式的研究提供新的思路和见解。
西北农林科技大学博士研究生李昭宏和硕士研究生李东维为该论文共同第一作者,杨若林教授为通讯作者。相关工作得到国家自然科学基金、陕西省人才项目和西北农林科技大学基金项目等资助。
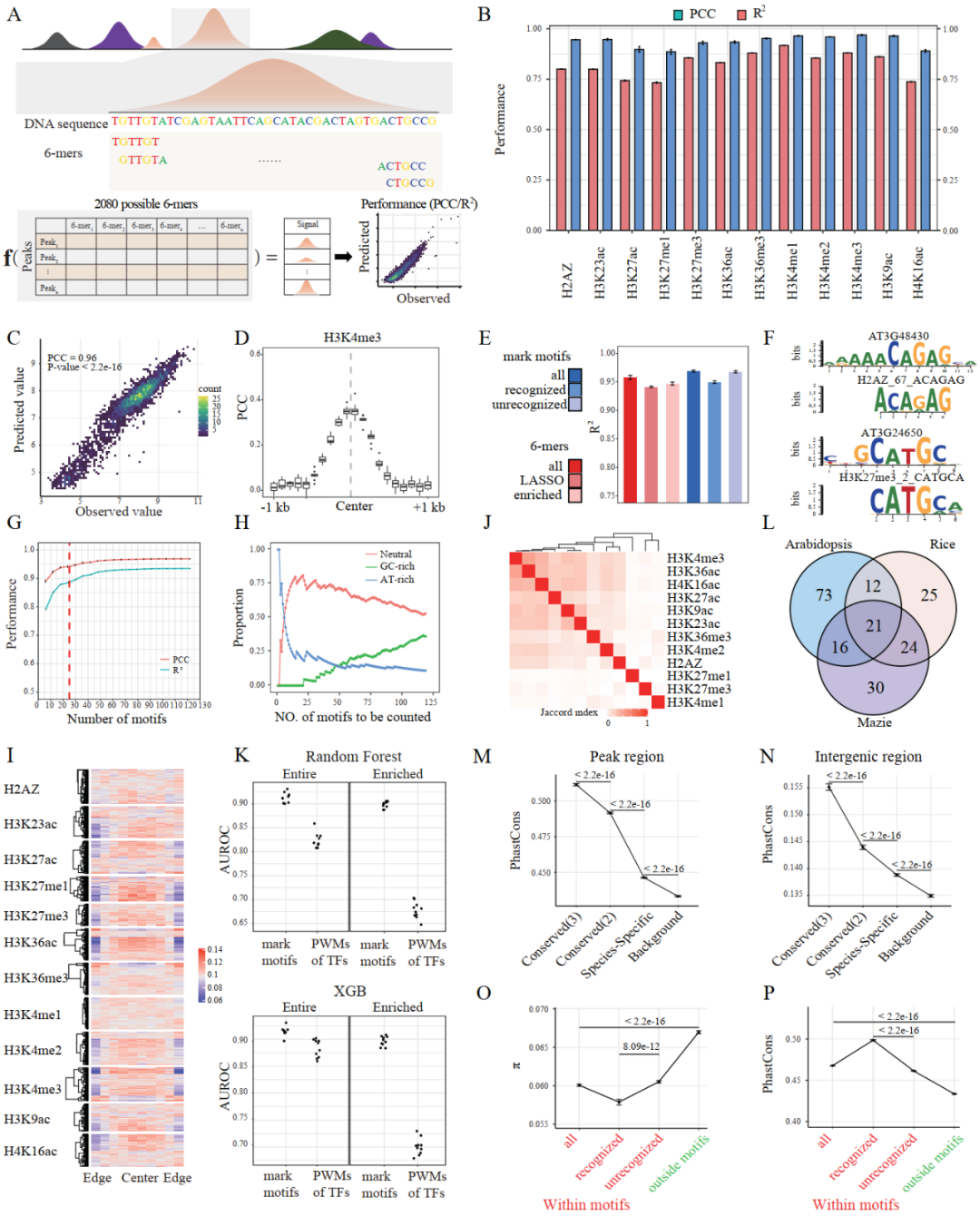
植物组蛋白修饰调控语法解析
A:所用模型示意图;B-E:不同序列特征的模型预测效果;F-J:mark motif的序列特征及其预测能力;K:不同序列特征在区分叶和根特异性H3K4me3修饰位点的表现;L:物种间mark motif的同源性;M-P:mark motif受到较强的功能约束。
文章链接:https://www.sciencedirect.com/science/article/pii/S1673852722001837
